FastKing gDNA Dispelling RT SuperMix
Funksjoner
■ Raskt: Ett trinn for å fullføre genomfjerning og effektiv revers transkripsjon innen 18 minutter ved bare å legge til maler.
■ Høy effektivitet: Omvendt transkriptase er modifisert med et hydrofobt motiv, med RT -effektivitet over 95%.
■ Enkel og enkel: Den eksklusive termosensitive DNase har rask effekt, høy effektivitet med kortere reaksjonstid og påvirker ikke cDNA.
Spesifikasjon
Type: Genmodifisert revers transkriptase, gDNase
Fremgangsmåter: Ett trinn (genomisk DNA-fjerning og RT)
RT -effektivitet: > 95%
Mal: 1 ng- 2 μg
Driftstid: ~ 18 min
Applikasjoner: Omvendt transkribert cDNA kan brukes i konvensjonell PCR, sanntids PCR, cDNA bibliotekskonstruksjon, SAGE (Serial Analysis of Gene Expression), primerforlengelse og andre konvensjonelle eksperimenter.
Alle produktene kan tilpasses for ODM/OEM. For detaljer,vennligst klikk på Tilpasset service (ODM/OEM)
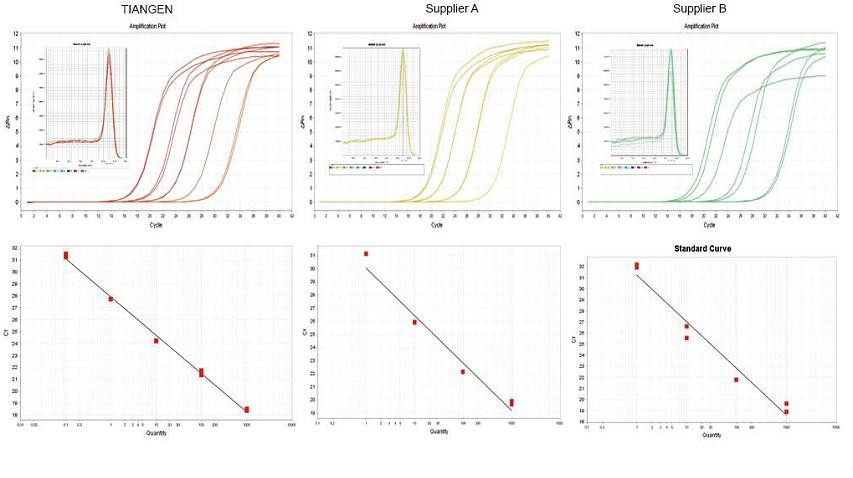 |
Eksperimentelt eksempel 1. cDNA ble syntetisert ved å bruke ett-trinns omvendte kvantitative reagenser fra TIANGEN FastKing gDNA Dispelling RT SuperMix, relevante produkter fra henholdsvis leverandør A og leverandør B. Oppdag RN5 -genet til mus ved bruk av TIANGEN Talent qPCR PreMix (SYBR Green), og forsterkningskurven, smeltekurven og standardkurven ble analysert. Resultatene viser at TIANGEN FastKing gDNA Dispelling RT SuperMix har den høyeste kvantitative Ct -verdien etter revers transkripsjon og utmerket stressmotstand, og har åpenbare fordeler for maler med høye urenhetsrester. |
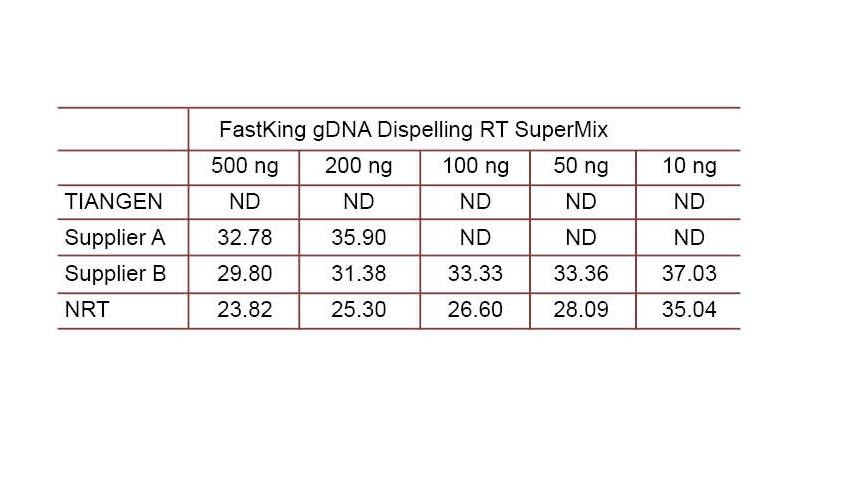 |
Eksperimentelt eksempel 2. cDNA ble syntetisert ved bruk av ett-trinns omvendte kvantitative reagenser fra TIANGEN FastKing gDNA Dispelling RT SuperMix, relevante produkter fra henholdsvis leverandør A og leverandør B. Oppdag humant HsG -gen ved bruk av TIANGEN Talent qPCR PreMix (SYBR Green), og legg manuelt til forskjellige konsentrasjoner av genomisk DNA for å oppdage evnen til fjerning av gDNA for forskjellige reagenser. CT -resultater viser at TIANGEN FastKing gDNA Dispelling RT SuperMix har utmerket evne til å fjerne genomisk DNA. Opptil 500 ng genomisk DNA -rest kan fjernes perfekt uten å påvirke resultatene. ND: Ikke oppdaget. NRT: Påvisning av blandingen uten revers transkripsjon. |
A-1 RNA nedbrytes
——Rens RNA av høy kvalitet uten forurensning. Materialet som RNA utvinnes fra skal være så friskt som mulig for å forhindre nedbrytning av RNA. Analyser RNA -integritet på denaturert gel før RT -reaksjon. Etter RNA -ekstraksjon, bør den lagres i 100% formamid. Hvis RNase -hemmer brukes, bør oppvarmingstemperaturen være <45 ° C, og pH bør være mindre enn 8,0, ellers vil inhibitoren frigjøre all bundet RNase. Videre bør RNase -hemmer tilsettes i løsninger som inneholder ≥ 0,8 mM DTT.
A-2 RNA inneholder hemmere av revers transkripsjonsreaksjoner
—— Reverse transkripsjonshemmere inkluderer SDS, EDTA, glyserol, natriumpyrofosfat, spermidin, formamid, guanidinsalt, etc. Bland kontroll -RNA med prøven, og sammenlign utbyttet med kontroll -RNA -reaksjonen for å kontrollere om det er en hemmer. Vask RNA -nedbør med 70% (v/v) etanol for å fjerne hemmere.
A-3 Utilstrekkelig gløding av primere som brukes til syntetisering av den første cDNA-strengen
—— Bestem at glødingstemperaturen er egnet for primerne som ble brukt i forsøket. For tilfeldige heksamerer anbefales det å holde temperaturen på 25 ° C i 10 minutter før reaksjonstemperaturen nås. For genspesifikke primere (GSP), prøv annen GSP, eller bytt til oligo (dT) eller tilfeldig heksamer.
A-4 Liten mengde start-RNA
—— Øk mengden RNA. For RNA -prøver mindre enn 50 ng, kan 0,1 μg til 0,5 μg acetyl BSA brukes i den første streng cDNA -syntesen
A-5 Målsekvensen uttrykkes ikke i de analyserte vevene.
- Prøv andre vev.
A-6 PCR-reaksjon mislykkes
—— For totrinns RT-PCR kan cDNA-malen i PCR-trinnet ikke overstige 1/5 av reaksjonsvolumet.
A-1 Ikke-spesifikk gløding av primere og maler
—— 3'-enden av primere bør ikke inneholde 2-3 dG eller dC. Bruk Genspesifikke primere i syntesen av første streng i stedet for tilfeldige primere eller oligo (dT). Bruk en høyere glødetemperatur i de første syklusene, og deretter en lavere glødetemperatur. Bruk Hot-start Taq DNA-polymerase for PCR for å forbedre spesifisiteten til reaksjonen.
A-2 Dårlig design av genspesifikke primere
——Følg de samme prinsippene for forsterkningsprimer -design.
A-3 RNA forurenset med genomisk DNA
-Behandle RNA med PCR-klasse DNase I. Sett opp en kontrollreaksjon uten revers transkripsjon for å oppdage DNA-forurensning.
A-4 Forming av primer dimer
—— Design primere uten komplementære sekvenser i 3' -enden.
A-5 For høy mg2+ konsentrasjon
—— Optimaliser Mg2+ konsentrasjon for hver mal- og primerkombinasjon
A-6 Forurenset med fremmed DNA
—— Bruk aerosolresistente tips og UDG-enzymer.
A-1 Innholdet i det første strengproduktet er for høyt
—— Reduser mengden av det første strengproduktet i det konvensjonelle PCR -reaksjonstrinnet.
A-2 For høy primermengde i PCR-reaksjon
—— Reduser primerinngang.
A-3 For mange sykluser
—— Optimaliser PCR -reaksjonsbetingelsene og reduser PCR -syklusnummeret.
A-4 For lav glødetemperatur
—— Øk glødetemperaturen for å forhindre uspesifikk initiering og forlengelse.
A-5 Ikke-spesifikk forsterkning av oligonukleotidfragmenter generert ved DNase-nedbrytning av DNA —— Ekstraher RNA av høy kvalitet for å forhindre DNA-forurensning.
RT-PCR er å reversere transkribering av RNA til cDNA, og deretter bruke det omvendte transkriberte cDNA som en mal for PCR-reaksjon for å forsterke målfragmentet. Velg enten tilfeldige primere, Oligo dT og genspesifikke primere i henhold til eksperimentets spesifikke forhold. Alle de ovennevnte primerne kan brukes for korte eukaryote celle -mRNA uten hårnålestruktur.
Tilfeldig primer: Passer for langt RNA med hårnålestruktur, så vel som alle slags RNA som rRNA, mRNA, tRNA, etc. De brukes hovedsakelig til RT-PCR-reaksjon av en enkelt mal.
Oligo dT: Egnet for RNA med PolyA -tailing (prokaryotisk RNA, eukaryotisk Oligo dT rRNA og tRNA har ikke PolyA -haler). Fordi Oligo dT er bundet til PolyA-halen, må kvaliteten på RNA-prøver være høy, og selv en liten nedbrytning vil i stor grad redusere mengden cDNA-syntese i full lengde.
Genspesifikk primer: Komplementær til mallsekvensen, egnet for situasjoner der målsekvensen er kjent.
Det er to måter:
1. Intern referansemetode: I teorien er cDNA DNA -fragmenter av forskjellige lengder, så resultatet av elektroforese er flekker. Hvis RNA -mengden er lav, vil ikke noe produkt vises ved elektroforese, men dette betyr ikke at intet produkt vil bli forsterket av PCR. Generelt kan intern referanse brukes til å oppdage cDNA. Hvis den interne referansen har resultater, kan kvaliteten på cDNA i utgangspunktet garanteres (i noen få tilfeller, hvis målgenfragmentet er for langt, kan det være unntak).
2. Hvis det er et kjent gen som er forsterket av denne malen, kan det verifiseres av primerne til dette genet. Forsterkningen av intern referanse betyr ikke nødvendigvis at det ikke er noe problem med cDNA. Fordi intern referanse har høy overflod i cDNA, er den lett å forsterke. Hvis cDNA er delvis degradert av forskjellige årsaker, sett fra sannsynlighetsperspektivet, vil PCR -resultater av målgener med lav overflod bli sterkt påvirket. Selv om intern referanse fortsatt er høy i overflod, vil forsterkningen sannsynligvis ikke bli påvirket.
Nedbrytes delvis av RNA. Oppdag integriteten og rens av RNA
RNA -innholdet til forskjellige arter kan være forskjellig, men generelt bør det ekstraherte totale RNA inneholde to klare 28S- og 18S -bånd i gelelektroforese, og lysstyrken til det tidligere båndet bør være dobbelt så høy som det sistnevnte. 5S -båndet indikerer at RNA er blitt degradert, og lysstyrken er proporsjonal med graden av nedbrytning. Den vellykkede forsterkningen av intern referanse betyr ikke at det ikke er noe problem med RNA, fordi den interne referansen er i stor mengde, RNA kan forsterkes så lenge nedbrytningen ikke er alvorlig. OD260/OD280forholdet mellom rent RNA målt ved spektrofotometer bør være mellom 1,9 og 2,1. En liten mengde protein urenhet i RNA vil redusere forholdet. Så lenge verdien ikke er for lav, vil RT ikke bli påvirket. Det som betyr mest for RT er RNA -integritet.
Forlengelsen av det interne referansegenet kan bare indikere at RT har lyktes, men det er ikke nødvendigvis relatert til kvaliteten på cDNA -strengen. Fordi de interne referansefragmentene generelt er små i størrelse og høy i uttrykk, er det lettere å lykkes med revers transkripsjon. Imidlertid varierer størrelsen og uttrykket til målgenet fra gen til gen. CDNA -kvaliteten kan ikke bare bedømmes etter intern referanse, spesielt for målfragmentene som er lengre enn 2 kb.
Noen prøver har komplekse sekundære strukturer, eller har et rikt GC -innhold, eller er dyrebare med lav overflod. I disse tilfellene bør passende revers transkriptase velges i henhold til størrelsen på målfragmentet og prøven. For RNA -maler med høyt GC -innhold og kompleks sekundær struktur er det vanskelig å åpne den sekundære strukturen ved lav temperatur, eller med vanlig revers transkriptase. For disse malene kan Quant Reverse Transcriptase velges, siden dens revers transkripsjonsytelse åpenbart er bedre enn M-MLV-seriens revers transkriptase, som kan reversere transkribering av forskjellige RNA-maler effektivt og transkribere RNA til cDNA første streng i maksimal grad. Når du bruker generelle revers transkriptasesett, kan 20 ul system bare effektivt reversere transkribering av 1 μg totalt RNA. Vær oppmerksom på settets maksimale RT -kapasitet. Hvis malen legges til i overkant, vil revers transkripsjon favorisere RNA med høy overflod. Derfor er det bedre å ikke overskride systemets maksimale kapasitet.
A-1 Bestem om RNA nedbrytes alvorlig og om RT er vellykket
Generelt er årsaken til svikt i intern referanseforsterkning ofte forårsaket av alvorlig RNA -nedbrytning. En annen mulig årsak er revers transkripsjonssvikt. Intern referanse kan ikke brukes som en standard for å bedømme kvaliteten på cDNA -enkeltstreng, men den kan brukes som en standard for å bedømme om revers transkripsjon er vellykket hvis det ikke er noe problem med RNA -kvaliteten. Det viktigste i omvendt transkripsjon er å opprettholde en konstant temperatur og et konstant reaksjonssystem for å forbedre reaksjonseffektiviteten.
A-2 Bestem om primerne for amplifikasjon av interne referansegener er pålitelige og om det er problemer med reagenser som brukes i PCR.
For relativ kvantifisering må RNA kvantifiseres før revers transkripsjon, noe som også kreves i mange revers transkripsjonssett, for eksempel kvantifiserer RNA -inngangen som 1 μg. Siden omvendt transkribert cDNA er en blandet løsning, inkludert RNA, oligo dT, enzym, dNTP, og til og med en liten DNA -rest, vil avvik forårsakes, så det er umulig å kvantifisere cDNA nøyaktig. Derfor er RNA -kvantifisering nødvendig. Med tanke på omvendt transkripsjonseffektivitet er den samme blant forskjellige prøver, bør mengden av oppnådd cDNA være den samme, og den kvantitative analysen kan vise sammenligning av ekspresjonsnivåer for forskjellige gener i samme mengde totalt RNA. Når du utfører relativ fluorescens kvantitativ PCR, er det ikke nødvendig med kvantitativt cDNA etter revers transkripsjon fordi det interne referansegenet kan fungere som referanse.
Det er hovedsakelig relatert til genene, og revers transkripsjon av langt fragment er ikke mulig for de fleste gener. For det første er effektiviteten av revers transkripsjon langt lavere enn for PCR. For det andre begrenser den GC -rike regionen og sekundære strukturen til mange gener både revers transkripsjon og PCR. Endelig er troverdigheten og forsterkningseffektiviteten til PCR vanskelig å garantere samtidig. I prosessen med revers transkripsjon kan ingen garantere å få langt fragment for gener med lav kopi, spesielt ved bruk av oligo dT. Når det gjelder 5 'UTR med mer GC, er det enda vanskeligere. Derfor er det fortsatt en rimelig metode for å reversere transkripsjon med tilfeldige primere, finne de naturlige spaltingsstedene i målfragmentet, forsterkes med segmenter og deretter utføre restriksjonsfordøyelsen og ligeringen. Generelt er det vanskelig å amplifisere fragmenter større enn 2 kb direkte, men det er ikke alltid umulig å oppnå: 1. Først av alt, garantere integriteten til RNA/mRNA, og TRIZOL -ekstraksjon foretrekkes. 2.M-MLV RT-PCR-sett kan brukes direkte. Forleng glødetid og øk syklusnummeret i forsterkningsprosessen riktig. Alternativt kan nestet PCR påføres, eller utføre en eller to reaksjoner først med passende forlenget denaturering og forlengelsestid før normal PCR -forsterkning, noe som kan bidra til å forlenge fragmenter. Vær oppmerksom på polymerasens troskap. 3. Long Taq kan brukes i PCR for å oppnå ideelle resultater. 4.For proteinuttrykk, bør high -fidelity polymerase påføres.
Det er to typer revers transkriptase som tilbys av TIANGEN: Quant/King RTase og TIANScript M-MLV. Hovedforskjellen mellom dem er inputmengden av maler. Quant er en unik revers transkriptase, som er forskjellig fra den vanlige M-MLV avledet fra Moloney murin leukemi virus. Quant er en ny høyeffektiv revers transkriptase rekombinant uttrykt av engineering Escherichia coli. Quant er egnet for å forsterke 50 ng-2 μg RNA med høy revers transkripsjonell aktivitet og høyt utbytte. Sammenlignet med vanlige MMLV eller AMV, er Quant største kjennetegn at den har en veldig sterk affinitet med RNA -maler og kan reversere transkripsjon av komplekse maler uten denaturering ved høy temperatur. For maler med høyere GC -innhold er omvendt effektivitet høyere. Imidlertid har denne revers transkriptase RNase H -aktivitet, noe som kan påvirke lengden på cDNA -produktet (egnet for <4,5 kb maler). For konvensjonell revers transkripsjon anbefales TIANScript MMLV revers transkriptase. Denne RTase er et modifisert enzym med svært svak RNase H -aktivitet, som er egnet for lang (> 5 kb) cDNA -syntese.
Ett-trinns omvendt transkripsjon og PCR-forsterkning fullføres i samme rør uten å åpne rørdekselet mellom cDNA-syntese og forsterkning, noe som er nyttig for å redusere forurensning. Siden alle oppnådde cDNA -prøver brukes til amplifikasjon, er sensitiviteten høyere, med minimum 0,01 pg totalt RNA. For vellykket ett-trinns RTPCR brukes genspesifikke primere generelt for å starte cDNA-syntese. To-trinns metoden, nemlig revers transkripsjon og PCR-amplifikasjon, utføres i to trinn. For det første utføres omvendt transkripsjon fra en RNA -mal for å oppnå cDNA, og det oppnådde cDNA utsettes for en eller flere forskjellige PCR -reaksjoner. To-trinns metoden kan bruke oligo (dT) eller tilfeldige primere for å styre syntesen av den første cDNA-strengen, og kan reversere transkribere all mRNA-informasjon fra en bestemt prøve.
Produktkategorier
HVORFOR VELGE OSS
Siden etableringen har fabrikken utviklet produkter i førsteklasses klasse med prinsippet
av kvalitet først. Våre produkter har fått et godt rykte i bransjen og er verdifulle blant nye og gamle kunder.